
| Microexon ID | Gm_13:31591358-31591372:+ |
| Species | Glycine max | Coordinates | 13:31591358..31591372 |
| Microexon Cluster ID | MEP45 |
| Size | 15 |
| Phase | 2 |
| Pfam Domain Motif | RPE65 |
| Structure of Microexon-tag (flanking exon, microexon, flanking exon sizes) | 47,15,46 |
| Microexon location in the Microexon-tag | 2 |
| Microexon-tag DNA Seq | AGRGTTGGKCCTAAYCCMAAGTTTGYYCCWGTKGCTGGATAYCAYTGGTTTGATGGAGATGGMATGATTCATGSYWTGCGYATYAAAGATGGAAAAGCWACWTATGTY |
| Logo of Microexon-tag DNA Seq | 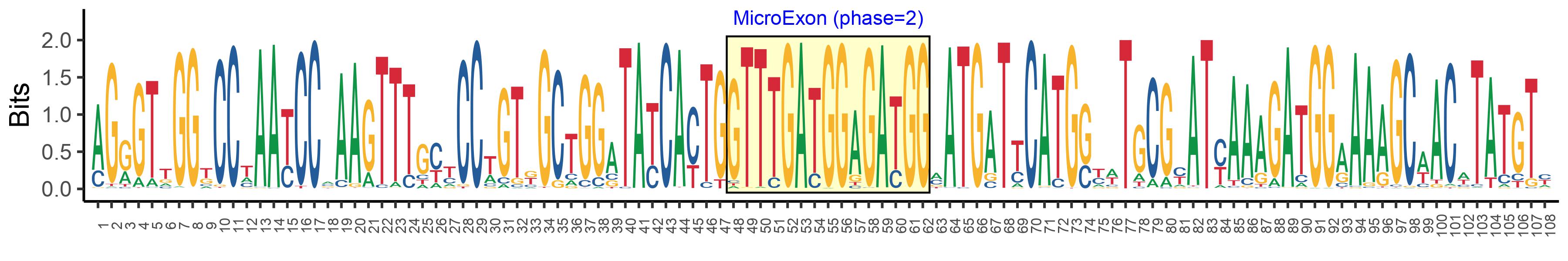 |
| Alignment of exons | 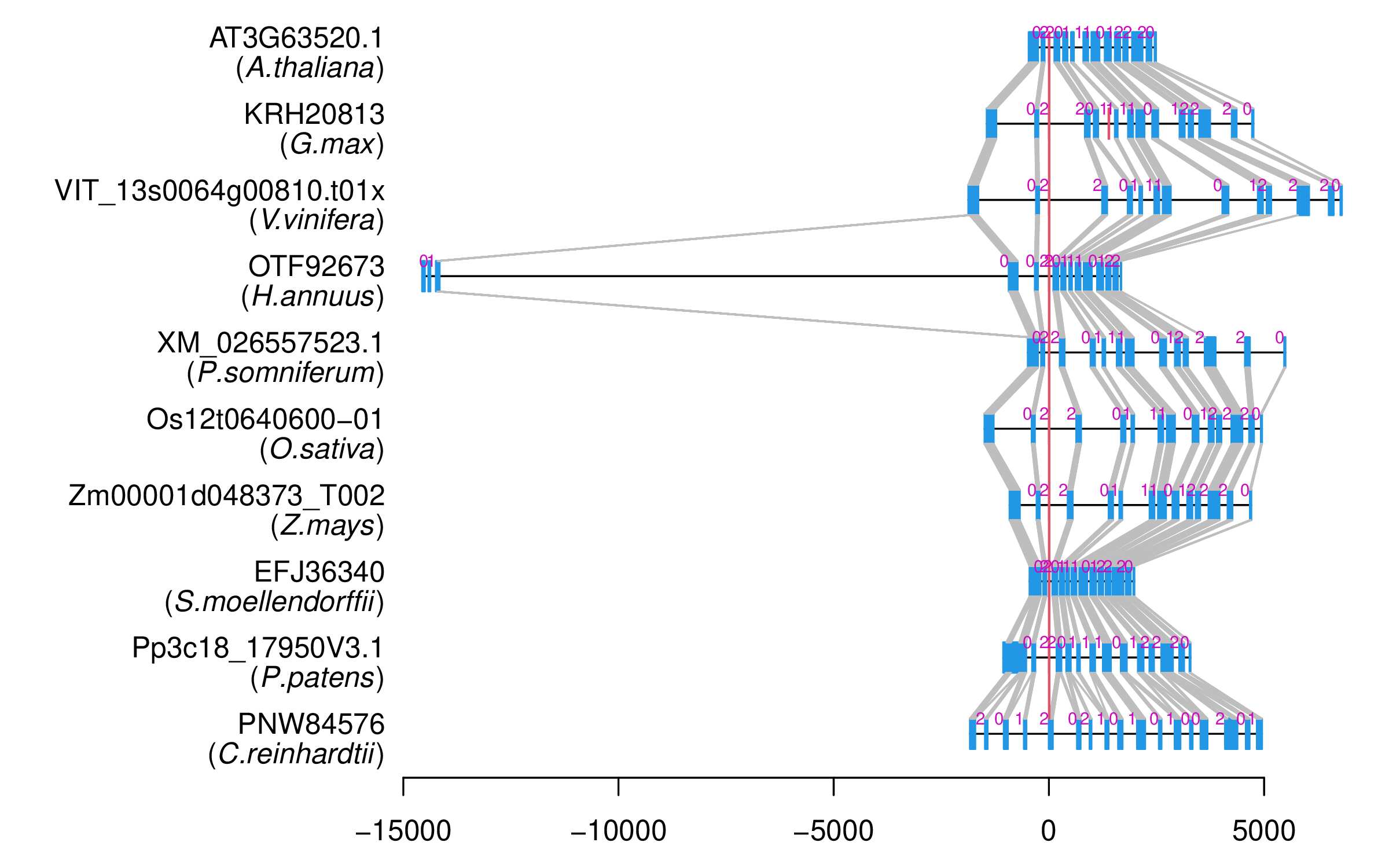 |
| Microexon DNA seq | GTTTGATGGAGATGG |
| Microexon Amino Acid seq | WFDGDG |
| Microexon-tag DNA Seq | AGAGTTGGGCCTAATCCGAAGTTTGCTCCTGTAGCTGGATATCACTGGTTTGATGGAGATGGAATGATTCATGGCTTGCGCATCAAAGATGGAAAAGCTACATATGTT |
| Microexon-tag Amino Acid Seq | RVGPNPKFAPVAGYHWFDGDGMIHGLRIKDGKATYV |
| Microexon-tag spanning region | 31591067-31592243 |
| Microexon-tag prediction score | 0.9899 |
| Overlapped with the annotated transcript (%) | 100 |
| New Transcript ID | KRH20813x |
| Reference Transcript ID | KRH20813 |
| Gene ID | GLYMA_13G202200 |
| Gene Name | NA |
| Transcript ID | KRH20813 |
| Protein ID | KRH20813 |
| Gene ID | GLYMA_13G202200 |
| Gene Name | NA |
| Pfam domain motif | RPE65 |
| Motif E-value | 1.1e-131 |
| Motif start | 55 |
| Motif end | 537 |
| Protein seq | >KRH20813 MGDDGKKISGEGGLVKVEPKPSNGFTSKVVDLLEKLVVKFLYDSSLPHHYLTGNFAPVSETPPTKDLPVKGYLPDCLNGE FVRVGPNPKFAPVAGYHWFDGDGMIHGLRIKDGKATYVSRFVRTSRLKQEEYFGGSKFMKIGDLKGLFGLLMVNIHMLRT KWKVLDASYGTGTESALHANTALVYHHGKLLALSEADKPYAIKVFEDGDLQTLGMLDYDKRLGHSFTAHPKVDPFTGEMF TFGYAHTPPYITYRVISKDGYMHDPVPITVSDPIMMHDFAITENYAIFLDLPLIFRPKEMVKNKTLIFSFDSTKKARFGV LPRYAKDEKLIRWFELPNCFIFHNANAWEEEDEVVLITCRLQNPNLDLVGGTAKEKLENFSNELYEMRFNMKTGEASQKK LSASAVDFPRVNESYTGRKQRYVYGTTLDSIAKVTGIIKFDLHAEPDNGKTKLEVGGNVQGLYDLGPGKYGSEAVYVPRV PGTDSEEDDGYLICFVHDENTGKSFVHVINAKTMSADPVAVVELPHRVPYGFHAFFVTEEQLQEQGKL* |
| CDS seq | >KRH20813 ATGGGGGATGATGGAAAGAAGATTAGTGGAGAGGGGGGGTTAGTGAAGGTTGAGCCAAAACCCAGCAATGGTTTCACCTC AAAAGTGGTTGATTTGTTGGAGAAATTGGTGGTGAAGTTCTTGTATGATTCTTCACTGCCCCATCACTACCTCACTGGTA ATTTTGCTCCCGTGAGTGAGACCCCTCCAACCAAGGACCTTCCTGTCAAAGGGTACCTTCCGGATTGCTTGAATGGGGAG TTTGTCAGAGTTGGGCCTAATCCGAAGTTTGCTCCTGTAGCTGGATATCACTGGTTTGATGGAGATGGAATGATTCATGG CTTGCGCATCAAAGATGGAAAAGCTACATATGTTTCCCGTTTTGTGAGAACTTCTCGCCTTAAACAAGAAGAATACTTTG GAGGCTCCAAATTTATGAAGATTGGAGATCTCAAAGGTCTATTCGGACTTTTAATGGTTAACATACATATGCTGAGAACT AAATGGAAAGTGCTGGATGCTTCTTATGGAACTGGAACAGAGAGTGCATTACATGCTAATACTGCTCTCGTATATCACCA TGGGAAACTTCTAGCACTCTCAGAAGCAGATAAACCCTATGCTATTAAAGTTTTTGAAGATGGTGATTTGCAGACACTTG GCATGCTAGATTATGACAAGAGATTGGGCCACTCCTTCACTGCTCATCCAAAAGTTGACCCATTTACTGGGGAGATGTTT ACATTTGGCTATGCGCATACACCACCATATATCACATACAGAGTAATTTCAAAGGATGGTTATATGCATGATCCTGTACC CATAACAGTATCAGATCCCATCATGATGCACGACTTTGCCATCACAGAGAATTATGCAATATTTTTGGATCTTCCTTTGA TTTTTAGGCCAAAGGAAATGGTGAAGAATAAGACACTGATATTCTCATTTGATTCAACCAAGAAAGCTCGTTTTGGTGTG CTACCTCGGTATGCTAAGGATGAAAAGCTTATTAGATGGTTTGAGTTACCCAACTGCTTCATATTCCACAATGCCAATGC TTGGGAGGAGGAAGATGAAGTTGTTCTGATCACATGCCGCCTTCAGAATCCAAATTTGGATTTGGTCGGTGGGACTGCCA AGGAAAAGCTTGAAAATTTCTCAAATGAGCTGTATGAAATGAGATTTAACATGAAAACGGGTGAAGCTTCTCAAAAGAAA CTATCAGCATCTGCTGTAGATTTTCCTAGGGTGAATGAGAGCTACACTGGAAGGAAGCAGCGATATGTATACGGAACCAC ATTAGACAGCATTGCAAAAGTTACTGGCATTATTAAATTTGATTTGCATGCTGAACCGGATAATGGAAAAACAAAACTTG AAGTAGGAGGAAATGTTCAGGGTCTCTATGACTTGGGACCAGGGAAGTATGGCTCGGAAGCTGTTTATGTCCCTCGTGTC CCTGGTACCGATTCTGAAGAAGATGATGGATACTTGATTTGTTTTGTACACGATGAAAATACCGGAAAATCATTTGTGCA TGTCATCAATGCAAAAACAATGTCAGCAGATCCTGTTGCAGTTGTCGAATTGCCGCATAGAGTTCCATATGGTTTCCATG CCTTCTTTGTGACAGAGGAACAACTGCAAGAGCAGGGTAAACTGTGA |
| Microexon DNA seq | GTTTGATGGAGATGG |
| Microexon Amino Acid seq | WFDGDG |
| Microexon-tag DNA Seq | AGAGTTGGGCCTAATCCGAAGTTTGCTCCTGTAGCTGGATATCACTGGTTTGATGGAGATGGAATGATTCATGGCTTGCGCATCAAAGATGGAAAAGCTACATATGTT |
| Microexon-tag Amino Acid seq | RVGPNPKFAPVAGYHWFDGDGMIHGLRIKDGKATYV |
| Transcript ID | KRH20814 |
| Gene ID | Gm.12466 |
| Gene Name | NA |
| Pfam domain motif | RPE65 |
| Motif E-value | 1.9e-132 |
| Motif start | 55 |
| Motif end | 532 |
| Protein seq | >KRH20814 MGDDGKKISGEGGLVKVEPKPSNGFTSKVVDLLEKLVVKFLYDSSLPHHYLTGNFAPVSETPPTKDLPVKGYLPDCLNGE FVRVGPNPKFAPVAGYHWFDGDGMIHGLRIKDGKATYVSRFVRTSRLKQEEYFGGSKFMKIGDLKGLFGLLMVNIHMLRT KWKVLDASYGTGTANTALVYHHGKLLALSEADKPYAIKVFEDGDLQTLGMLDYDKRLGHSFTAHPKVDPFTGEMFTFGYA HTPPYITYRVISKDGYMHDPVPITVSDPIMMHDFAITENYAIFLDLPLIFRPKEMVKNKTLIFSFDSTKKARFGVLPRYA KDEKLIRWFELPNCFIFHNANAWEEEDEVVLITCRLQNPNLDLVGGTAKEKLENFSNELYEMRFNMKTGEASQKKLSASA VDFPRVNESYTGRKQRYVYGTTLDSIAKVTGIIKFDLHAEPDNGKTKLEVGGNVQGLYDLGPGKYGSEAVYVPRVPGTDS EEDDGYLICFVHDENTGKSFVHVINAKTMSADPVAVVELPHRVPYGFHAFFVTEEQLQEQGKL* |
| CDS seq | >KRH20814 ATGGGGGATGATGGAAAGAAGATTAGTGGAGAGGGGGGGTTAGTGAAGGTTGAGCCAAAACCCAGCAATGGTTTCACCTC AAAAGTGGTTGATTTGTTGGAGAAATTGGTGGTGAAGTTCTTGTATGATTCTTCACTGCCCCATCACTACCTCACTGGTA ATTTTGCTCCCGTGAGTGAGACCCCTCCAACCAAGGACCTTCCTGTCAAAGGGTACCTTCCGGATTGCTTGAATGGGGAG TTTGTCAGAGTTGGGCCTAATCCGAAGTTTGCTCCTGTAGCTGGATATCACTGGTTTGATGGAGATGGAATGATTCATGG CTTGCGCATCAAAGATGGAAAAGCTACATATGTTTCCCGTTTTGTGAGAACTTCTCGCCTTAAACAAGAAGAATACTTTG GAGGCTCCAAATTTATGAAGATTGGAGATCTCAAAGGTCTATTCGGACTTTTAATGGTTAACATACATATGCTGAGAACT AAATGGAAAGTGCTGGATGCTTCTTATGGAACTGGAACAGCTAATACTGCTCTCGTATATCACCATGGGAAACTTCTAGC ACTCTCAGAAGCAGATAAACCCTATGCTATTAAAGTTTTTGAAGATGGTGATTTGCAGACACTTGGCATGCTAGATTATG ACAAGAGATTGGGCCACTCCTTCACTGCTCATCCAAAAGTTGACCCATTTACTGGGGAGATGTTTACATTTGGCTATGCG CATACACCACCATATATCACATACAGAGTAATTTCAAAGGATGGTTATATGCATGATCCTGTACCCATAACAGTATCAGA TCCCATCATGATGCACGACTTTGCCATCACAGAGAATTATGCAATATTTTTGGATCTTCCTTTGATTTTTAGGCCAAAGG AAATGGTGAAGAATAAGACACTGATATTCTCATTTGATTCAACCAAGAAAGCTCGTTTTGGTGTGCTACCTCGGTATGCT AAGGATGAAAAGCTTATTAGATGGTTTGAGTTACCCAACTGCTTCATATTCCACAATGCCAATGCTTGGGAGGAGGAAGA TGAAGTTGTTCTGATCACATGCCGCCTTCAGAATCCAAATTTGGATTTGGTCGGTGGGACTGCCAAGGAAAAGCTTGAAA ATTTCTCAAATGAGCTGTATGAAATGAGATTTAACATGAAAACGGGTGAAGCTTCTCAAAAGAAACTATCAGCATCTGCT GTAGATTTTCCTAGGGTGAATGAGAGCTACACTGGAAGGAAGCAGCGATATGTATACGGAACCACATTAGACAGCATTGC AAAAGTTACTGGCATTATTAAATTTGATTTGCATGCTGAACCGGATAATGGAAAAACAAAACTTGAAGTAGGAGGAAATG TTCAGGGTCTCTATGACTTGGGACCAGGGAAGTATGGCTCGGAAGCTGTTTATGTCCCTCGTGTCCCTGGTACCGATTCT GAAGAAGATGATGGATACTTGATTTGTTTTGTACACGATGAAAATACCGGAAAATCATTTGTGCATGTCATCAATGCAAA AACAATGTCAGCAGATCCTGTTGCAGTTGTCGAATTGCCGCATAGAGTTCCATATGGTTTCCATGCCTTCTTTGTGACAG AGGAACAACTGCAAGAGCAGGGTAAACTGTGA |